
應用方向:
在本研究中,高光譜成像技術被應用于茶樹扦插苗的生長監測與建模,展現出在農業育種與智能管理中的廣闊應用前景。通過對葉片和嫩梢的高光譜反射數據采集,結合深度學習可以在不破壞樣本的前提下實現茶苗地上部和根系生物量的快速、精準預測,解決根系難以直接測量的問題,為替代傳統耗時且破壞性的稱重方法提供了有效途徑。因此,高光譜技術不僅能夠為茶樹優良品種的篩選和扦插苗的生長監測提供可靠手段,還可為苗圃精細化管理和智能化育苗奠定技術基礎。
背景:
茶樹是全球重要的經濟作物,具有龐大的消費市場,但茶苗的繁育與生長受到不良氣候條件等因素的限制,導致茶樹扦插苗生長緩慢、成本高昂,從而制約了優良品種的推廣與產業化水平。扦插苗的地上部和根系生物量是衡量其生長發育質量的重要指標,因此,如何快速、準確地監測其生長過程對于提高成活率和苗木管理水平具有重要意義。
傳統的人工稱重方法在測定茶樹扦插苗生物量時具有破壞性且效率低,難以滿足高通量、無損檢測的需求。近年來,高光譜成像技術因其能夠同時獲取樣品的結構和化學信息,被廣泛應用于作物性狀監測。已有研究表明,光譜信息與植物的生理、生化特征存在緊密聯系,可用于反映植株的養分含量、激素水平及生理狀態。結合機器學習和深度學習的方法,能夠進一步提升高光譜數據的建模精度與穩定性。
因此,該研究提出利用高光譜成像技術結合深度學習方法,對茶樹扦插苗的地上部和根系生物量進行快速、無損預測,以解決傳統方法耗時、破壞性強的問題,為茶樹優良品種的高效篩選和精細化育苗管理提供新的技術途徑和數據支撐。
作者信息:丁兆堂;山東省農業科學院
期刊來源:Scientia Horticulturae
研究內容
本研究旨在利用高光譜成像技術結合深度學習模型,實現對茶樹扦插苗地上部和根系生物量的快速、無損與精準預測,以解決傳統人工稱重方法破壞性強、效率低的問題。研究過程中,首先采集不同生長階段茶樹扦插苗葉片與嫩梢的高光譜反射數據,并對光譜數據進行預處理,以去除噪聲和散射效應并增強光譜與生理指標的相關性。隨后,提取關鍵光譜特征波段,減少冗余信息并突出與生物量密切相關的變量。在建模環節,利用 Mask R-CNN 網絡對扦插苗進行圖像與光譜特征融合,構建CNN-GRU生物量預測模型。研究建立了一種高效可靠的檢測方法,為茶樹優良品種的篩選、扦插苗的生長監測以及智能化育苗管理提供了新的技術途徑與數據支撐。
實驗設計
茶樹扦插苗選用了三個品種:‘玉金香(YJX)’、‘中白1號(ZB)’和‘中茗6號(ZM)’。為了獲取不同生長期的茶扦插苗,實驗每25天進行一次,共進行了10次。每次試驗1個穴盤(含32株扦插苗),共收獲扦插苗960株(3個品種× 32株扦插苗× 10個試驗)。
為了測定扦插苗的生長情況,對960條扦插苗的枝條和根系進行破壞性分析,并采集高光譜數據,將枝條和根系置于105 ℃烘箱中20 min,然后將烘箱溫度調至90 ℃干燥至恒重,最后用電子秤記錄重量
本研究采用了高光譜成像技術來監測茶扦插苗從扦插到成長為幼苗過程中生物量的變化(見圖1a)。高光譜成像采集系統包括成像光譜相機(Gaia field pro-v10,江蘇雙利合譜技術有限公司),四個鹵素燈、電腦、校正白板、黑色背景。高光譜相機所拍攝圖像的光譜范圍在可見-近紅外波段(391-1010 nm),光譜范圍為1101 × 960像素,可測量360個波段的光譜反射率。為了避免光譜相機內部暗電流的影響,提高高光譜圖像的信噪比,對獲取的原始高光譜圖像進行黑白白色校正。利用高光譜相機采集了960株扦插苗的高光譜數據,每幅高光譜圖像包含8株扦插苗作為一個模型樣本,得到包括120個高光譜圖像的總數據集。通過結合成熟葉片和莖葉的光譜信息以及深度學習和機器學習算法,對莖葉和根系的生物量進行了評估(見圖1b)。
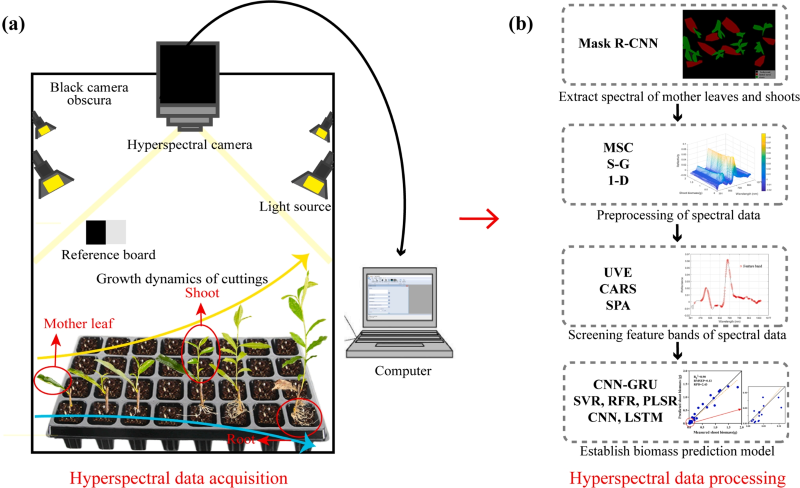
圖1. 圖像采集與流程圖的結合。(a) 圖像采集 (b) 數據處理流程圖
研究方法
為了增加模型訓練的數據量,120張圖像通過兩種不同的數據增強方法進行處理:旋轉(90°、180°和270°)和翻轉(水平和垂直),圖像數量增加到720張。這些圖像通過Labelme軟件進行手動標注。首先,對成熟葉片和莖葉區域分別用不同顏色的標簽進行標注和分類,其中綠色代表莖葉,紅色代表成熟葉片。未標注的區域被視為背景。圖2展示了成熟葉片和莖葉的標注圖像。隨后,將這些標注圖像輸入到Mask R-CNN中進行訓練。

圖2. 標注圖像 (a) 標注成熟葉片和莖葉的圖像;(b) 標注完成的成熟葉片和莖葉圖像
Mask R-CNN網絡被用于獲取莖葉和成熟葉片的面積。圖3展示了Mask R-CNN網絡的結構圖。Mask R-CNN 網絡主要分為五個結構:Backbone、區域建議網絡(RPN)、感興趣區域對齊(ROI Align)、邊框回歸(Box Regression)以及分類與掩膜(Classification and Mask)。通過5折交叉驗證將標記的圖像劃分為訓練集和測試集。所采用的學習率為0.001,Epoch為20,Batch大小為1。
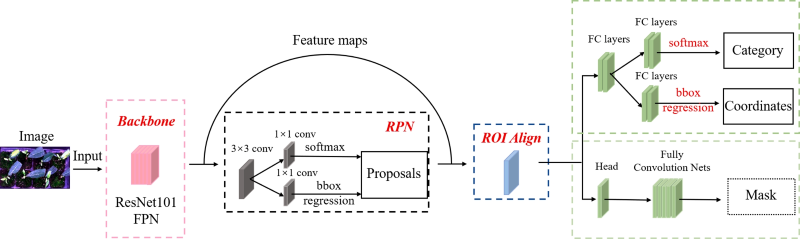
圖3. Mask R-CNN的結構圖。
由于高光譜采集儀器及環境因素的影響,在成熟葉片和嫩梢的光譜中存在散射效應、隨機噪聲和系統噪聲。因此,本研究對其光譜數據進行了 MSC、S-G 和一階導數(1-D)預處理。另外為了減少數據計算量并提高后續建模的準確性,本研究采用了連續投影算法(SPA)、競爭自適應重加權采樣算法(CARS)和不確定性變量消除算法(UVE)來選擇具有代表性的光譜波段作為特征波段。
在模型的建立方面,構建了卷積神經網絡與門控循環單元(CNN-GRU)網絡模型和傳統機器學習模型。CNN-GRU模型網絡結構如圖4所示。首先,為了更好地提取數據的底層特征,使用CNN來提取數據特征。然后,數據被輸入到一個5×5的濾波器中進行卷積。經過4次卷積、平均池化、序列擴展和展平后,數據被輸入到GRU網絡中。在這里,一維光譜數據與莖葉和根系生物量數據被GRU網絡結合,用于回歸預測。此外,在CNN網絡中,步長為1,填充為“same"(填充值由算法根據卷積核大小內部計算),輸入數據通道為1。經過3次門控循環后,預測數據被輸入到全連接層,并通過回歸器輸出。
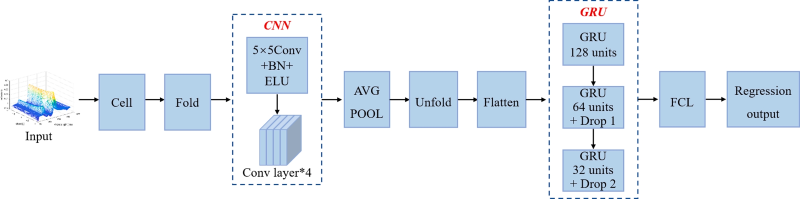
圖4. CNN-GRU的結構圖。
為了進一步驗證CNN-GRU網絡的性能,使用了SVR、RFR和PLSR三種機器學習方法和CNN、LSTM兩種深度學習方法作為對比模型,CNN和LSTM網絡的層數均設置為16層。為了防止模型的過擬合,在訓練過程中確定了超參數,支持向量回歸機的核函數為多項式核函數,RFR的樹的數量為200,PLSR的隱變量為18。在本研究中,用6種方法分別建立了以地上部、成熟葉和地上部、根系生物量為指標的回歸模型。
為了進一步保證算法的準確性,本研究采用五重交叉驗證,將數據集分成五部分,依次取其中4個作為訓練數據,1個作為測試數據,重復五次,然后對結果進行平均,為了更準確地評價回歸模型的性能,使用決定系數(R2)、均方根誤差(RMSE)、歸一化均方根誤差(NRMSE)和相對百分比偏差(RPD)來評價模型的性能。為了評估Mask R-CNN模型的光譜信息提取性能,使用精度,召回率和F1得分來評估模型的性能。
結果
通過實驗室方法測定了茶扦插苗的莖葉和根系生物量。結果顯示,三種品種的扦插苗莖葉萌發時間相同,均在第25天開始萌發。然而,中茗6號(ZM)的生根時間最早,從第125天開始生根;中茗6號的莖葉和根系生長量最大,莖葉生長量約為1.7克,根系生長量約為0.6克。在200–225天期間,莖葉和根系的生長速度最快;中白1號(ZB)的生根時間最晚,從第175天開始生根;玉金香(YJX)的莖葉和根系生長量最小,莖葉生長量約為0.8克,根系生長量約為0.1克。
對成熟葉片和莖葉的分割結果進行了比較和分析,結果顯示,Mask R-CNN能夠以高精度分割成熟葉片和莖葉。其中,成熟葉片光譜的提取效果優于莖葉光譜,提取精度達到97.8%。莖葉光譜的提取精度為91.5%。成熟葉片和莖葉光譜的提取精度均超過90%。因此,Mask R-CNN模型能夠準確且高效地從茶扦插苗圖像中提取成熟葉片和莖葉的光譜信息。
對Mask R-CNN模型提取的成熟葉片和枝條原始光譜分別采用MSC、1-D和S-G算法進行預處理,如圖5所示,與原始光譜相比,MSC、1-D和S-G聯合預處理后光譜曲線的波峰和波谷更加突出,提高了光譜的分辨率和靈敏度,有利于提高后期建立回歸模型的準確性。
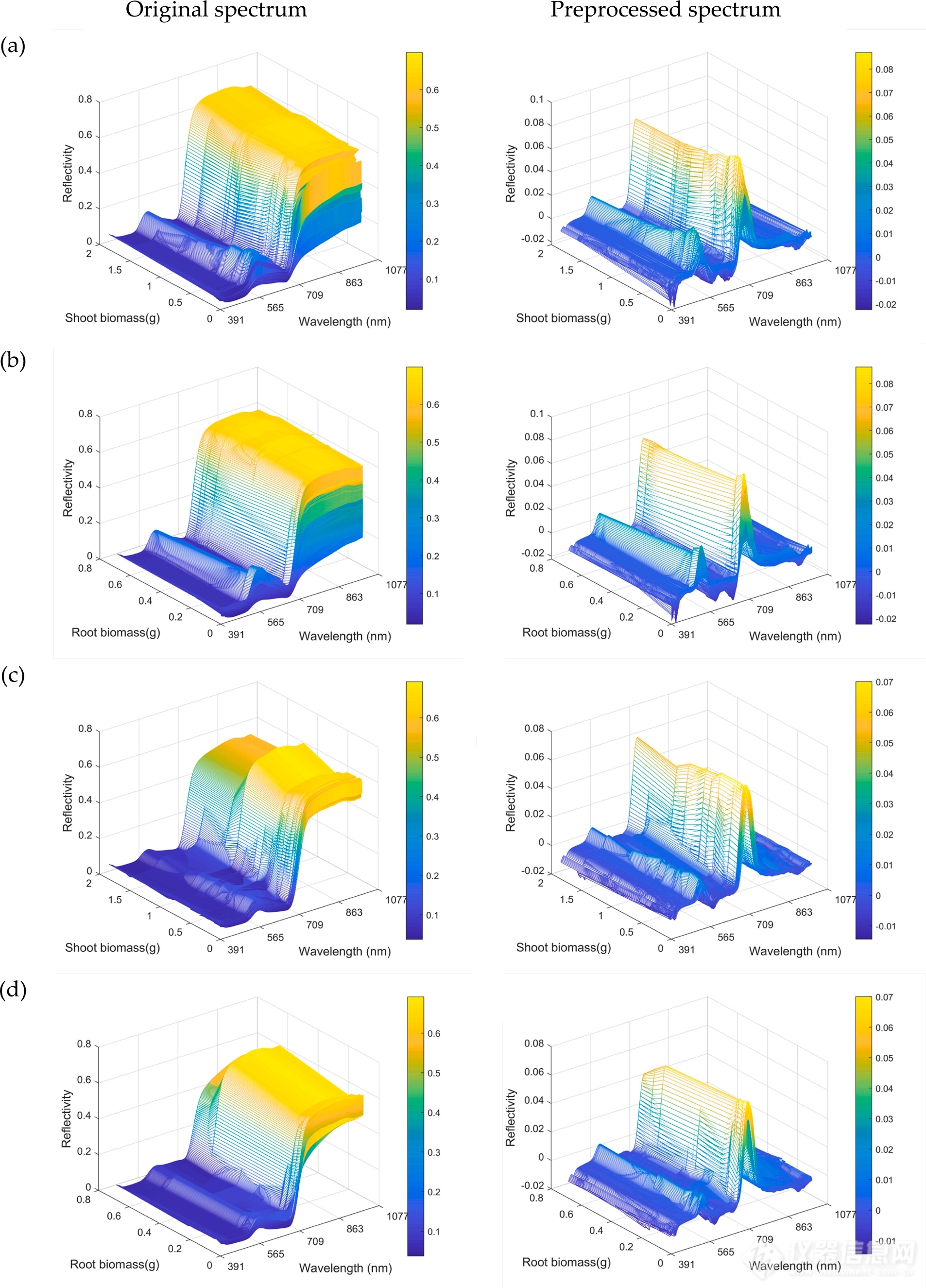
圖5. 原始光譜與MSC、1D和S-G預處理后的光譜對比。(a) 莖葉光譜 + 莖葉生物量;(b) 莖葉光譜 + 根系生物量;(c) 成熟葉片光譜 + 莖葉生物量;(d) 成熟葉片光譜 + 根系生物量。
為了消除無關波段對模型準確性的影響,在基于莖葉光譜選擇莖葉生物量特征波段的方法中,UVE選擇的特征波段數量最多,達到212個波長,而SPA選擇的特征波段數量最少,僅為8個波長;在基于莖葉光譜選擇根系生物量特征波段的方法中,UVE選擇的特征波段數量最多,為135個波長,SPA選擇的特征波段數量最少,為6個波長;在基于成熟葉片光譜選擇莖葉生物量特征波段的方法中,UVE選擇的特征波段數量最多,為69個波長,SPA選擇的特征波段數量最少,為14個波長;在基于成熟葉片光譜選擇根系生物量特征波段的方法中,UVE選擇的特征波段數量最多,為90個波長,SPA選擇的特征波段數量最少,為17個波長。
在基于莖葉光譜評估莖葉生物量時,UVE算法的建模效果優于CARS和SPA,UVE+CNN-GRU提供了最佳的估算模型(Rp2=0.90,RMSEP=0.12,RPD=2.43)。CARS算法的建模效果較差,CARS+PLSR模型的效果最差(Rp2=0.50,RMSEP=0.32,RPD=1.36)。
在基于成熟葉片光譜評估莖葉生物量時,UVE算法的建模效果優于CARS和SPA,UVE+CNN-GRU提供了最佳的估算模型(Rp2=0.78,RMSEP=0.16,RPD=2.13)。SPA算法的建模效果較差,SPA+PLSR模型的效果最差(Rp2=0.48,RMSEP=0.29,RPD=1.00)。在基于成熟葉片光譜評估根系生物量時,SPA算法的建模效果優于UVE和CARS,SPA+LSTM提供了最佳的估算模型(Rp2=0.65,RMSEP=0.05,RPD=1.67)。CARS算法的建模效果較差,CARS+PLSR模型的效果最差(Rp2=0.39,RMSEP=0.10,RPD=1.22)。圖6展示了四種最佳估算模型的預測值與實際值的散點圖。
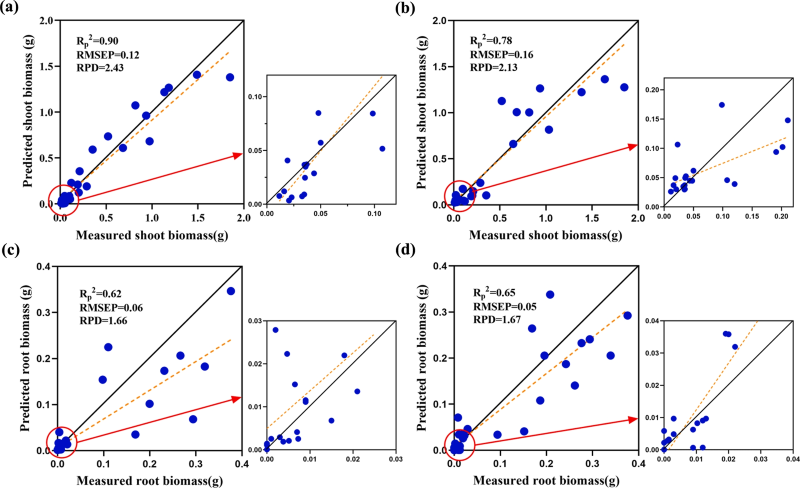
圖6. 四種最佳估算模型的預測值與實際值的散點圖。(a) 莖葉光譜 + UVE + CNN-GRU;(b) 成熟葉片光譜 + UVE + CNN-GRU;(c) 莖葉光譜 + SPA + CNN;(d) 成熟葉片光譜 + SPA + LSTM。
結論
本研究提出了一種利用高光譜成像技術監測茶扦插苗莖葉生長和根系生長的方法。首先,通過Mask R-CNN提取茶扦插苗成熟葉片的光譜和莖葉的光譜。隨后,利用MSC、S-G濾波和1-D對光譜進行預處理,并通過UVE、CARS和SPA篩選特征波段。最后,采用CNN-GRU網絡構建莖葉和根系生物量的預測模型。研究結果表明:(1)Mask R-CNN能精確提取成熟葉片(精確率=97.8%)和嫩梢(精確率=91.5%)的光譜特征;(2)通過UVE方法篩選獲得的嫩梢(212個)和根系(105個)生物量特征波段,較CARS和SPA方法更具豐富性;(3)基于嫩梢光譜構建的UVE+CNN-GRU模型(Rp2=0.90,RMSEP=0.12,RPD=2.43)對嫩梢生物量的估算效果*優,表明該模型預測結果可靠,與實際值誤差較小;(4)基于成熟葉片光譜構建的SPA+LSTM模型(Rp2=0.65,RMSEP=0.05,RPD=1.67)對根系生物量的估算效果*佳,證明該模型可用于茶樹扦插苗根系狀況評估,為根系生長監測提供了有效手段。